

Aplicaciones de la metagenómica en el estudio de alimentos fermentados: una perspectiva desde la química de alimentos
Applications of metagenomics in the study of fermented foods: a food chemistry perspective
Azucena Máximo-Olguín1,a, Jesús Guadalupe Pérez-Flores1,b, Laura García-Curiel1,c,*, Emmanuel Pérez-Escalante1,d, Elizabeth Contreras-López1,e, Judith Jaimez-Ordaz1,f, Alma Elizabeth Cruz-Guerrero2,g
1 Universidad Autónoma del Estado de Hidalgo, Mineral de la Reforma, México.
2 Universidad Autónoma Metropolitana, Ciudad de México, México.
a Est.,
![]() azucenamaximoolguin@gmail.com,
azucenamaximoolguin@gmail.com, ![]() https://orcid.org/0009-0007-3218-144X
https://orcid.org/0009-0007-3218-144X
b Dr., ![]() jesus_perez@uaeh.edu.mx,
jesus_perez@uaeh.edu.mx, ![]() https://orcid.org/0000-0002-9654-3469
https://orcid.org/0000-0002-9654-3469
c Dra., ![]() laura.garcia@uaeh.edu.mx,
laura.garcia@uaeh.edu.mx, ![]() https://orcid.org/0000-0001-8961-2852
https://orcid.org/0000-0001-8961-2852
d Dr., ![]() emmanuel_perez@uaeh.edu.mx,
emmanuel_perez@uaeh.edu.mx, ![]() https://orcid.org/0000-0002-4268-9753
https://orcid.org/0000-0002-4268-9753
a Dra.,
![]() elizac@uaeh.edu.mx,
elizac@uaeh.edu.mx, ![]() https://orcid.org/0000-0002-9678-1264
https://orcid.org/0000-0002-9678-1264
a Dra.,
![]() jjaimez@uaeh.edu.mx,
jjaimez@uaeh.edu.mx, ![]() https://orcid.org/0000-0001-6655-0759
https://orcid.org/0000-0001-6655-0759
a Dra, ![]() aec@xanum.uam.mx,
aec@xanum.uam.mx, ![]() https://orcid.org/0000-0002-9686-2267
https://orcid.org/0000-0002-9686-2267
* Autor de Correspondencia: Tel. +52 1 771 152 3666
http://doi.org/10.25127/riagrop.20254.1132
La creciente demanda de alimentos seguros, funcionales y de alta calidad ha impulsado la aplicación de herramientas metagenómicas en los campos de la microbiología y la química alimentaria. Este artículo analizó sus aplicaciones en alimentos fermentados, con énfasis en su utilidad para caracterizar comunidades microbianas y su relación con los cambios químicos, sensoriales y funcionales, a fin de evaluar su impacto en la calidad, la inocuidad alimentaria y el desarrollo de nuevos productos. La metodología comprendió una revisión temática de estudios publicados entre 2015 y 2025, que abordaron técnicas metagenómicas, diversidad microbiana en alimentos fermentados, compuestos bioactivos y aplicaciones emergentes. Los resultados mostraron que la metagenómica facilita la detección de microorganismos, la predicción de vías metabólicas y la identificación de genes relacionados con deterioro, patogenicidad y metabolitos funcionales. Sus aplicaciones incluyen la evaluación del riesgo microbiológico, la optimización de fermentaciones y el diseño de cultivos iniciadores o probióticos. No obstante, persisten limitaciones asociadas al costo, la infraestructura, la complejidad bioinformática y la escasa representación microbiana en bases de datos. Se proponen enfoques integrativos con química analítica y ciencias ómicas para superarlas. En conjunto, la metagenómica se perfila como una herramienta poderosa para innovar en el diseño, la inocuidad y la funcionalidad de alimentos fermentados.
Palabras claves: Fermentación, inocuidad alimentaria, metabolitos, metagenómica, microbiota.
Abstract
The demand for safe, functional, and high-quality foods has promoted the use of metagenomic tools in microbiology and food chemistry. This article analyzed their applications in fermented foods, emphasizing their usefulness in characterizing microbial communities and their relationship with chemical, sensory, and functional changes, in order to assess their impact on quality, food safety, and the development of new products. The methodology involved a thematic review of studies published between 2015 and 2025, addressing metagenomic techniques, microbial diversity in fermented foods, bioactive compounds, and emerging applications. The results showed that metagenomics facilitates the detection of microorganisms, the prediction of metabolic pathways, and the identification of genes related to spoilage, pathogenicity, and functional metabolites. Its applications include microbiological risk assessment, fermentation process optimization, and the design of starter cultures or probiotic formulations. However, limitations remain regarding cost, infrastructure, bioinformatic complexity, and the underrepresentation of food-specific microorganisms in reference databases. Integrative approaches combining metagenomics with analytical chemistry and other omics sciences are proposed to address these challenges. Overall, metagenomics emerges as a powerful tool to drive innovation in the design, safety, and functionality of fermented foods.
Keywords: Fermentation, food safety, metabolites, metagenomics, microbiota.
1. INTRODUCCIÓN
La metagenómica es un campo de estudio con enfoque interdisciplinar que permite analizar el material genético directamente a partir de muestras ambientales, sin necesidad de aislar ni cultivar los microorganismos presentes. Este enfoque ha ampliado las posibilidades de la microbiología al facilitar la identificación y caracterización de comunidades microbianas en su entorno natural, incluyendo especies que no pueden cultivarse en condiciones de laboratorio (Chiriac & Murariu, 2021; Parras-Moltó & Aguirre De Cárcer, 2020). La implementación de tecnologías de secuenciación masiva, en particular el enfoque metagenómico tipo shotgun, ha sido relevante para el desarrollo del campo, ya que permite realizar análisis taxonómicos, funcionales y reconstrucciones genómicas directamente a partir de muestras microbianas complejas (Lin & Liao, 2016; Miller et al., 2018; Yue et al., 2020).
Este enfoque se ha aplicado en diversas disciplinas, entre ellas la microbiología y la química de alimentos. En estos contextos, la metagenómica permite caracterizar las comunidades microbianas que habitan las matrices alimentarias, evaluar su composición, funcionalidad metabólica y potencial patógeno, y vincular estos aspectos con la calidad química, sensorial y nutricional de los productos alimenticios (Beck et al., 2024; Yasir et al., 2023). También se ha aplicado en el estudio de alimentos fermentados, donde los perfiles microbianos influyen directamente en el desarrollo de compuestos aromáticos, metabolitos bioactivos y atributos sensoriales deseables (Srinivas et al., 2022; Wikandari et al., 2021).
Además, esta tecnología ha demostrado ser útil en la vigilancia de la inocuidad alimentaria. Entre sus aplicaciones destacan la detección de genes asociados con resistencia a antibióticos (resistoma), la identificación de microorganismos patógenos en alimentos listos para consumo y el rastreo de contaminaciones a lo largo de las cadenas productivas (Bloomfield et al., 2025; Eckstrom & Barlow, 2019; Wang et al., 2025). En el estudio del deterioro alimentario, la metagenómica ha permitido describir con mayor precisión los cambios en las comunidades microbianas que conducen a la pérdida de calidad o inocuidad (Das et al., 2023; Huang et al., 2017).
La fermentación es otro proceso donde la metagenómica ha generado aportes significativos. El análisis de consorcios microbianos en fermentaciones tradicionales ha permitido identificar microorganismos predominantes y caracterizar funciones metabólicas asociadas a la estabilidad del producto, el desarrollo del sabor, la textura y ciertos efectos funcionales (Gautam et al., 2024; Phiri et al., 2019; Zabat et al., 2018). Estos conocimientos han facilitado el desarrollo de cultivos iniciadores dirigidos y el diseño de alimentos con propiedades tecnológicas o nutricionales específicas.
A partir de lo anterior, este trabajo tuvo como objetivo analizar, mediante una revisión de literatura científica publicada entre 2015 y 2025, las aplicaciones de la metagenómica en el estudio de alimentos fermentados, considerando su contribución al conocimiento de las comunidades microbianas y su relación con los cambios químicos, sensoriales y funcionales del alimento, con el propósito de identificar sus alcances en calidad, inocuidad alimentaria y desarrollo de alimentos funcionales.
2. METAGENÓMICA APLICADA: FUNDAMENTOS, TÉCNICAS Y BENEFICIOS
2.1. Extracción de ADN
Con el fin de evitar sesgos y pérdidas de información es necesario obtener ADN metagenómico de alta calidad. Este proceso se ve dificultado por la diversidad estructural de las paredes celulares microbianas, lo que requiere protocolos robustos que garanticen la lisis eficiente en distintos tipos celulares. La aplicación de protocolos optimizados ha permitido mejorar la recuperación de ADN sin contaminación, condición necesaria para aplicaciones posteriores como la amplificación génica y la secuenciación (Hassan et al., 2018; Verma et al., 2017).
En estudios basados en genes marcadores, como el 16S rRNA, la PCR permite amplificar regiones específicas para su posterior análisis taxonómico. La elección de las regiones variables del gen marcador, así como de los pares de cebadores utilizados, puede influir en los resultados del análisis, ya que diferentes combinaciones pueden alterar la composición microbiana detectada (Rintala et al., 2017). Además de su uso en taxonomía, la PCR sirve como control de calidad del ADN extraído, permitiendo validar la idoneidad de las muestras para análisis metagenómicos (Shamim et al., 2017).
2.3. Plataformas de secuenciación
Las NGS han transformado el estudio de microbiomas al permitir un análisis detallado y de alto rendimiento del material genético. Estas plataformas procesan grandes volúmenes de secuencias en paralelo, lo que reduce significativamente el tiempo y los costos del análisis. Comparaciones entre distintos sistemas han evidenciado variaciones en velocidad, precisión y profundidad de lectura, factores que influyen en la elección de la tecnología según los objetivos del estudio (Allali et al., 2017). También se ha subrayado la necesidad de aplicar controles in silico adecuados (como conjuntos de referencia y simulaciones negativas) para evaluar y calibrar los algoritmos de clasificación taxonómica, con el fin de reducir falsos positivos y garantizar una identificación más confiable (Tennant et al., 2017).
2.4. Análisis bioinformático
El procesamiento e interpretación de los datos generados por secuenciación requieren herramientas bioinformáticas capaces de manejar grandes volúmenes de información. Existen pipelines especializados para análisis de datos de amplicones y metagenomas tipo shotgun. Algunos, como NG-Tax, han sido validados por su precisión en la asignación taxonómica para datos de 16S rRNA (Ramiro-Garcia et al., 2018). Otros, enfocados en agrupamiento genómico (binning) y visualización, permiten explorar interacciones ecológicas entre miembros de la comunidad microbiana (Sedlar et al., 2017).
La integración adecuada de estas técnicas, desde la extracción y secuenciación hasta el análisis computacional, binning y visualización es determinante para la calidad y utilidad de los resultados metagenómicos. La estandarización metodológica sigue siendo un objetivo en desarrollo, necesario para mejorar la reproducibilidad y aplicabilidad de los estudios en microbiología ambiental y de alimentos.
3. QUÍMICA DE LOS ALIMENTOS Y SU RELACIÓN CON LA MICROBIOTA
3.1. Cambios químicos inducidos por microorganismos
Durante el procesamiento y la fermentación de alimentos, los microorganismos transforman diversos sustratos en metabolitos que alteran la composición química del producto. Estas transformaciones generan compuestos con efectos sobre la estabilidad, el sabor y el valor nutricional. Entre los metabolitos más frecuentes se encuentran los ácidos orgánicos, los péptidos bioactivos y los compuestos volátiles.
Los ácidos orgánicos, como el ácido láctico, reducen el pH del alimento, lo que inhibe el crecimiento de microorganismos de deterioro y prolonga su vida útil. Además, su presencia intensifica notas de acidez que contribuyen al perfil sensorial del producto (Bourdichon et al., 2021). Este tipo de transformación es común en fermentaciones de productos lácteos, vegetales o cárnicos.
La hidrólisis de proteínas inducida por enzimas microbianas libera péptidos de bajo peso molecular con potencial bioactivo. Algunos de estos compuestos presentan propiedades antioxidantes o antihipertensivas, lo que añade un valor funcional a los alimentos fermentados (Bourdichon et al., 2021). La liberación de estos péptidos depende de la cepa microbiana y de las condiciones de fermentación.
Durante el metabolismo microbiano también se generan compuestos volátiles como ésteres, alcoholes y aldehídos. Estos contribuyen al aroma del producto final y pueden llegar a determinar su aceptabilidad. Su formación está influenciada por el tipo de microorganismo, el sustrato y las condiciones del proceso (Hu et al., 2022). La incorporación de cultivos iniciadores permite orientar la producción de estos metabolitos hacia perfiles deseados. Además, esta estrategia puede ser útil en esquemas de bioconservación como alternativa a los conservadores químicos (Rus‐Fernández & Fuentes, 2025).
3.2. Metabolitos secundarios microbianos y calidad alimentaria
Los metabolitos secundarios generados por la microbiota durante la fermentación cumplen funciones que van más allá del metabolismo primario, influyendo en la calidad, la vida útil y las propiedades sensoriales de los alimentos. Entre estos se encuentran bacteriocinas, exopolisacáridos (EPS) y compuestos aromáticos.
Las bacteriocinas son péptidos antimicrobianos producidos por bacterias ácido lácticas (BAL) que inhiben microorganismos patógenos o alterantes. Se han aplicado de forma exitosa en carnes, lácteos y otros alimentos frescos, donde pueden sustituir conservadores sintéticos sin alterar las propiedades sensoriales (Efendi et al., 2024; Rasheed et al., 2021).
Los EPS mejoran la textura, viscosidad y estabilidad de alimentos fermentados. Además, pueden actuar como agentes protectores frente a la deshidratación o como soportes para la liberación controlada de compuestos funcionales. Su producción ha sido asociada con mejoras en la aceptabilidad del producto (Akacha et al., 2023; Qiao et al., 2022).
Los compuestos aromáticos derivados de la fermentación determinan el carácter sensorial del alimento. Su producción se asocia con la actividad metabólica de levaduras y bacterias bajo condiciones específicas. En fermentaciones tradicionales como la del Baijiu, la sucesión microbiana ha sido relacionada con perfiles aromáticos complejos (Ban et al., 2024; Miao et al., 2023).
El uso conjunto de metabolitos microbianos con compuestos naturales como polifenoles ha sido propuesto como estrategia de conservación que respete las demandas del consumidor actual. Sin embargo, la variabilidad en la síntesis de estos metabolitos sugiere la necesidad de enfoques dirigidos para su control y optimización (Dutta et al., 2022; Yu et al., 2023).
3.3. Influencia de la fermentación sobre la composición química
La fermentación modifica la composición química de alimentos al transformar macronutrientes y micronutrientes mediante la actividad metabólica de los microorganismos. Estos cambios afectan la funcionalidad tecnológica y nutricional de los productos.
En el caso de las proteínas, la acción enzimática durante la fermentación incrementa su digestibilidad y libera péptidos bioactivos. Esta transformación puede mejorar la calidad proteica y conferir efectos fisiológicos positivos (Tamang et al., 2016).
Respecto a los carbohidratos, la fermentación degrada polímeros complejos a azúcares más simples, lo que facilita la disponibilidad energética y estimula el crecimiento de microbiota beneficiosa. La producción de ácidos orgánicos como resultado de este metabolismo favorece la estabilidad y acidez del alimento (Fan et al., 2023; Raghuvanshi et al., 2019).
En relación con los lípidos, se ha reportado que ciertas bacterias lácticas pueden modificar el perfil de ácidos grasos e incluso producir compuestos como el ácido linoleico conjugado (CLA), con potencial efecto bioactivo (Taylor et al., 2020).
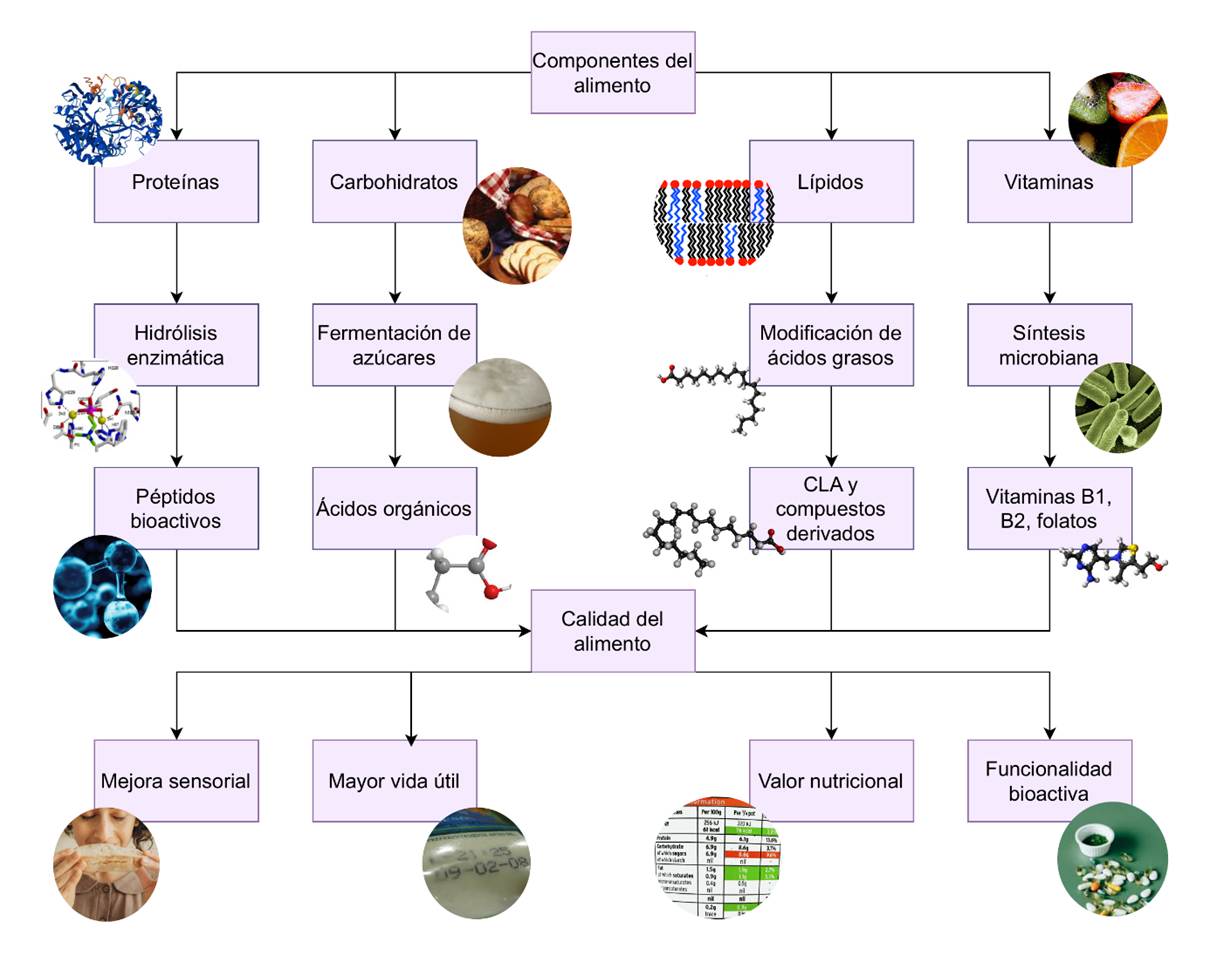
Figura 1. Interacción entre microbiota y compuestos químicos del alimento.
La fermentación también puede incrementar el contenido vitamínico de los alimentos. Las vitaminas del complejo B, como tiamina, riboflavina y ácido fólico, suelen sintetizarse en concentraciones mayores durante el proceso, como se ha documentado en fermentaciones de cereales y bebidas tradicionales (Chileshe et al., 2020; Tsafrakidou et al., 2020).
La Figura 1 muestra cómo las rutas metabólicas microbianas convergen en la formación de compuestos funcionales que influyen en las propiedades del alimento. Esta interacción depende del tipo de nutriente, la microbiota presente y las condiciones del entorno. Complementariamente, la Tabla 1 resume los compuestos modificados, los microorganismos involucrados y sus efectos en alimentos fermentados.
La actividad metabólica microbiana transforma proteínas, carbohidratos, lípidos y precursores vitamínicos en metabolitos funcionales que afectan la estabilidad, el perfil sensorial y el valor nutricional del alimento.
Tabla 1. Cambios químicos inducidos por microorganismos y su relación con la calidad alimentaria
|
Tipo de compuesto afectado |
Microorganismos implicados |
Cambios inducidos y efectos sobre el alimento |
Referencias |
|
Ácidos orgánicos |
BAL (e.g. Lactobacillus, Lactococcus, Leuconostoc), bacterias acéticas |
Disminución del pH, inhibición de patógenos, mejora de estabilidad y perfil sensorial |
(Bourdichon et al., 2021) |
|
Péptidos bioactivos |
Bacterias proteolíticas lácticas y hongos filamentosos |
Liberación de péptidos con efectos antioxidantes, antihipertensivos, antimicrobianos y moduladores del sistema inmune |
(Bourdichon et al., 2021; Tamang et al., 2016) |
|
Compuestos volátiles |
Levaduras (Saccharomyces, Kluyveromyces, Candida) y bacterias fermentativas |
Producción de ésteres, alcoholes y aldehídos y CO₂ que determinan el aroma, sabor y textura en productos fermentados |
(Ban et al., 2024; Hu et al., 2022; Miao et al., 2023) |
|
Bacteriocinas |
BAL (e.g. Lactobacillus, Pediococcus, Enterococcus) |
Inhibición de microorganismos alterantes, sustitución de conservadores sintéticos, mejora de la inocuidad alimentaria |
(Efendi et al., 2024; Rasheed et al., 2021) |
|
Exopolisacáridos (EPS) |
BAL y Bifidobacterium |
Mejora de textura, viscosidad, estabilidad y aceptabilidad sensorial, protección de probióticos |
(Akacha et al., 2023; Qiao et al., 2022) |
|
Carbohidratos |
Bacterias fermentativas (lácticas, Bifidobacterium, Bacteroides) y levaduras |
Hidrólisis de polisacáridos, mayor biodisponibilidad energética, formación de ácidos orgánicos y compuestos prebióticos |
(Fan et al., 2023; Raghuvanshi et al., 2019) |
|
Lípidos |
BAL, Propionibacterium freudenreichii, hongos filamentosos |
Modificación del perfil lipídico, producción de ácido linoleico conjugado (CLA), mejora del valor nutricional y sensorial |
(Taylor et al., 2020) |
|
Vitaminas (B1, B2, folatos) |
BAL, Propionibacterium, levaduras y hongos |
Incremento del contenido vitamínico (especialmente B12, folatos, riboflavina) durante fermentación de cereales, vegetales y bebidas |
(Chileshe et al., 2020; Tsafrakidou et al., 2020) |
|
Fitoquímicos / antioxidantes |
BAL y hongos filamentosos |
Liberación o biotransformación de compuestos fenólicos y antioxidantes, mejora del potencial bioactivo de alimentos vegetales |
(Bourdichon et al., 2021) |
4. RELEVANCIA DE LA METAGENÓMICA EN LA FERMENTACIÓN, CALIDAD Y FUNCIONALIDAD ALIMENTARIA
4.1. Fermentación y alimentos tradicionales
La metagenómica ha ampliado el conocimiento sobre las comunidades microbianas involucradas en la fermentación de alimentos tradicionales como yogur, kimchi, queso, kombucha y masa madre. Al prescindir del cultivo previo, esta herramienta permite analizar la diversidad microbiana y su potencial funcional con mayor resolución.
Estudios recientes han revelado la especificidad microbiana de distintos productos fermentados a escala global, identificando perfiles únicos en función del alimento, la región y las condiciones de fermentación (Du et al., 2023; Whon et al., 2021). En masa madre, por ejemplo, se ha demostrado que la estabilidad y el desempeño funcional de las comunidades microbianas responden a la composición de especies dominantes y sus interacciones (Calabrese et al., 2022). En fermentaciones como huangjiu, la integración de enfoques meta-ómicos ha permitido vincular géneros como Saccharopolyspora con funciones en el proceso (Liu et al., 2023).
Los datos metagenómicos también han contribuido a entender el papel de microorganismos no iniciadores en productos como queso y kimchi, donde especies subdominantes influyen en el perfil sensorial y en los beneficios potenciales para la salud (Leeuwendaal et al., 2022; Walsh et al., 2020). En el caso de alimentos fermentados tradicionales como el tempeh o el yucha, se han identificado especies dominantes como Lactobacillus, Leuconostoc y Weissella, asociadas con metabolitos bioactivos y propiedades conservantes (Pangastuti et al., 2019; Zhang et al., 2016).
La capacidad de esta tecnología para detectar especies funcionales ha fortalecido el vínculo entre la diversidad microbiana y la calidad del alimento. En fermentaciones de cacao, vinagre y kéfir, se han identificado taxones clave como Acetobacter pasteurianus, Lactobacillus plantarum y Lactobacillus kefiri, cuya actividad incide en la acidificación, la formación de compuestos volátiles y la producción de compuestos bioactivos (Agyirifo et al., 2019; Han et al., 2024; Rosales-Valdívia et al., 2024; Sumarmono et al., 2023).
Además de caracterizar la microbiota, los enfoques metagenómicos permiten predecir rutas metabólicas implicadas en la síntesis de vitaminas, péptidos bioactivos y otros compuestos con potencial funcional, ampliando el conocimiento sobre los posibles efectos benéficos de los alimentos fermentados (Yasir et al., 2023).
4.2. Calidad e inocuidad alimentaria
La metagenómica ha demostrado ser una estrategia robusta para la detección de patógenos y microorganismos de deterioro en productos alimentarios crudos y procesados. Al permitir el análisis del ADN microbiano sin cultivo previo, esta técnica ofrece una visión más completa de la microbiota presente, mejorando los sistemas de monitoreo en inocuidad alimentaria.
El uso combinado de metagenómica y métodos tradicionales ha optimizado la detección de patógenos como Listeria monocytogenes y Escherichia coli en frutas, verduras y carnes (Quek et al., 2025). En estudios de brotes, el análisis metagenómico a nivel de cepa ha permitido rastrear la fuente de Salmonella, aportando una resolución superior para la identificación y trazabilidad de agentes etiológicos (Buytaers, Saltykova, Mattheus, et al., 2021).
La secuenciación tipo shotgun se ha utilizado con éxito para detectar genes de resistencia antimicrobiana y patógenos como Clostridium botulinum y Staphylococcus aureus en alimentos procesados, lo que extiende las aplicaciones de esta técnica al ámbito de la vigilancia genómica (Lee et al., 2023). Además, enfoques como el metabarcoding y la secuenciación de lectura larga han mejorado la sensibilidad para detectar microorganismos deteriorantes y patógenos, ampliando el rango de identificación (Grützke et al., 2019).
Los estudios también han documentado el papel de géneros como Bacillus y Pseudomonas fragi en la alteración de frutas y productos refrigerados. La caracterización genómica ha permitido asociar estos microorganismos con rutas metabólicas relacionadas con el deterioro, lo que favorece el diseño de estrategias preventivas (De Filippis et al., 2019; Pant & Shashidhar, 2023).
El uso de la metagenómica como herramienta de monitoreo ha sido propuesto para evaluar procesos de deterioro bioquímico vinculados a contaminación y almacenamiento. Por ejemplo, se ha utilizado para detectar hongos productores de micotoxinas y revelar especies micotoxigénicas no identificadas por métodos convencionales (Ottesen et al., 2024). Además, el seguimiento de comunidades microbianas en almacenamiento ha mostrado ser útil para detectar cambios ecológicos asociados con pérdida de calidad (Bloomfield et al., 2025).
Este enfoque ha sido particularmente relevante en la identificación temprana de patógenos zoonóticos en matrices complejas como la leche cruda (Grützke et al., 2021), así como en investigaciones de brotes alimentarios, donde ha reducido significativamente los tiempos de respuesta en comparación con los métodos de cultivo (Buytaers, Saltykova, Denayer, et al., 2021; Saltykova et al., 2020). Finalmente, su integración con marcos regulatorios ha sido planteada como una estrategia para mejorar la trazabilidad y gestión del riesgo microbiológico en alimentos (Koutsoumanis et al., 2019; Kovac, 2019).
4.3. Desarrollo de alimentos funcionales
La metagenómica ha facilitado la identificación de microorganismos con propiedades benéficas y la caracterización de metabolitos asociados a beneficios para la salud, como péptidos bioactivos, probióticos y prebióticos. Estos hallazgos han contribuido al diseño y monitoreo de alimentos funcionales, particularmente aquellos basados en procesos fermentativos.
Análisis genómicos a gran escala han vinculado bacterias lácticas presentes en alimentos fermentados con componentes del microbioma intestinal humano, revelando su diversidad y potencial probiótico (Pasolli et al., 2020). Esta información ha permitido seleccionar cepas con funciones específicas para su incorporación en productos alimentarios. A través de la metagenómica funcional también se han identificado compuestos bioactivos con aplicaciones en salud, lo cual amplía el repertorio de ingredientes funcionales (Coughlan et al., 2015).
La caracterización precisa de cepas utilizadas en suplementos probióticos ha sido mejorada mediante secuenciación 16S rRNA, lo que garantiza su autenticidad y eficacia (Dioso et al., 2020). Comparaciones entre métodos dependientes del cultivo y análisis metagenómicos han demostrado que estos últimos ofrecen una visión más completa del perfil microbiano presente en formulaciones comerciales (Mazzantini et al., 2021).
El papel de los prebióticos en la modulación del microbioma también ha sido explorado mediante estudios metagenómicos. Se ha observado que su inclusión en la dieta puede favorecer la actividad probiótica mediante la selección de comunidades microbianas beneficiosas y la producción de metabolitos funcionales (Pineda-Quiroga et al., 2019). Esta sinergia ha reforzado la formulación de productos con propiedades funcionales dirigidas al intestino.
Las fermentaciones de alimentos tradicionales han permitido descubrir cepas autóctonas con potencial probiótico, como en vinos de arroz filipinos, donde se han identificado bacterias con propiedades funcionales prometedoras (Sanico & Medina, 2021). Esta estrategia amplía las posibilidades de innovación a partir de la biodiversidad microbiana local. En productos cárnicos fermentados, la incorporación de cepas seleccionadas también ha demostrado ser eficaz para mejorar beneficios funcionales (Munekata et al., 2020).
Además, la identificación de genes relacionados con la producción de péptidos bioactivos ha reforzado el interés en explorar alimentos fermentados como vehículos para compuestos con efectos antihipertensivos o antioxidantes (Lugli et al., 2022).
El uso de herramientas metagenómicas en la investigación y desarrollo de alimentos funcionales abarca distintas estrategias, desde la identificación de cepas probióticas hasta la mejora de matrices fermentadas mediante la modulación del microbioma o la obtención de metabolitos funcionales.
5. LIMITACIONES Y DESAFÍOS DE LA METAGENÓMICA EN ALIMENTOS
La implementación de tecnologías metagenómicas en el análisis de alimentos ha enfrentado diversas limitaciones técnicas, económicas y metodológicas. Estas limitaciones abarcan tanto los costos asociados con la infraestructura y el análisis de datos, como los retos en la interpretación funcional de los perfiles microbianos, especialmente en entornos con recursos limitados.
5.1. Costos y accesibilidad tecnológica
Pese a la disminución progresiva en los costos de secuenciación, el análisis metagenómico continúa siendo económicamente restrictivo para muchas instituciones, particularmente en regiones con infraestructura científica limitada. Plataformas como Oxford Nanopore o Pacific Biosciences han contribuido a mejorar el rendimiento de secuenciación y reducir los costos por muestra; sin embargo, el análisis metagenómico integral implica costos adicionales derivados de la preparación de bibliotecas, mantenimiento de equipos especializados y disponibilidad de personal capacitado (Muñoz-Martinez et al., 2025; Stewart et al., 2018; Tourlousse et al., 2021).
Este panorama se agrava en contextos de bajos recursos, donde el acceso a equipos de secuenciación y herramientas de análisis computacional es escaso. A pesar de que las soluciones basadas en la nube ofrecen alternativas accesibles para el procesamiento bioinformático, persisten barreras relacionadas con la capacitación técnica necesaria para interpretar conjuntos de datos complejos (Kalantar et al., 2020; Sun et al., 2023). En consecuencia, muchas iniciativas de vigilancia alimentaria o epidemiológica en estos entornos enfrentan obstáculos estructurales que limitan el aprovechamiento de la metagenómica.
5.2. Complejidad del análisis de datos y bioinformática
El procesamiento de datos metagenómicos genera desafíos asociados a la carga computacional, la disponibilidad de bases de datos de referencia adecuadas y la interpretación funcional de las comunidades microbianas. La cantidad de lecturas cortas generadas por las plataformas de secuenciación requiere herramientas específicas para ensamblaje, asignación taxonómica y binning, procesos que demandan recursos de cómputo avanzados y experiencia en bioinformática (Parrello et al., 2021; Qayyum et al., 2024; Tamames & Puente-Sánchez, 2019).
Las bases de datos actuales presentan una representación limitada de microorganismos no cultivables o propios de matrices alimentarias complejas, lo que restringe la precisión en la anotación funcional (Arango-Argoty et al., 2016; Oliveira et al., 2015). Esto se traduce en dificultades para inferir correctamente el papel ecológico o tecnológico de determinados taxones en alimentos fermentados, perecederos o mínimamente procesados (Santiago-Rodriguez et al., 2016; Zorrilla et al., 2021).
La interpretación de la diversidad y función microbiana se ve además afectada por la variabilidad entre muestras y la redundancia funcional de los taxones identificados. Dificultades en la preparación de muestras, como la presencia de ADN huésped o la heterogeneidad entre matrices, pueden sesgar los resultados y dificultar su comparación entre estudios (Bloomfield et al., 2025; Pasolli et al., 2019). Estas complejidades metodológicas limitan la capacidad para establecer asociaciones sólidas entre microbiota, procesos tecnológicos y atributos del producto final.
5.3. Necesidad de integración interdisciplinaria
El aprovechamiento pleno de la metagenómica en el contexto alimentario requiere su integración con disciplinas como la química analítica, la biotecnología y la microbiología aplicada. Esta convergencia permite fortalecer el vínculo entre la estructura genética de las comunidades microbianas y su impacto real en la calidad e inocuidad de los alimentos.
En este sentido, la combinación de secuenciación metagenómica con técnicas como qPCR, citometría de flujo o espectrometría de masas puede mejorar la detección de patógenos y metabolitos bioactivos, así como validar las funciones inferidas a partir de datos genómicos (Quek et al., 2025; Sim et al., 2024). Además, el uso de estrategias como la quasimetagenómica ha facilitado el subtipado de microorganismos relevantes como Listeria monocytogenes o Salmonella, complementando los análisis de riesgo microbiológico (Hyeon et al., 2018).
La aplicación de la metagenómica en alimentos continúa enfrentando obstáculos que condicionan su implementación efectiva y su aprovechamiento en contextos productivos o de vigilancia. Estos desafíos están vinculados a cuestiones técnicas o económicas y a la necesidad de integrar enfoques interdisciplinarios que permitan traducir los datos genéticos en soluciones concretas para la industria alimentaria.
6. CONCLUSIONES
El análisis de la literatura evidenció que la metagenómica es una herramienta prometedora para estudiar comunidades microbianas en alimentos fermentados, especialmente cuando los métodos tradicionales son limitados. Ha permitido ampliar el conocimiento sobre la diversidad microbiana, sus funciones y su papel en la producción de metabolitos con efectos tecnológicos y funcionales. También se destacó su utilidad para mejorar la inocuidad mediante la detección de patógenos y genes de resistencia. Sin embargo, persisten desafíos como los altos costos, la complejidad bioinformática y la baja representación de ciertos microorganismos en bases de datos. Se recomienda avanzar hacia enfoques integradores con otras técnicas ómicas, plataformas más accesibles y metodologías estandarizadas para potenciar su aplicación en el diseño y optimización de alimentos fermentados seguros y funcionales.
Declaración de intereses
El manuscrito fue preparado y revisado con la participación de todos los autores, quienes declaramos que no existe ningún conflicto de intereses que ponga en riesgo la validez de los resultados presentados.
Agradecimientos
Los autores expresan su agradecimiento al Sistema Nacional de Investigadoras e Investigadores (SNII) de la Secretaría de Ciencia, Humanidades, Tecnología e Innovación (SECIHTI) y al programa Estancias Posdoctorales por México de la SECIHTI por la beca otorgada a L. García-Curiel (CVU 625969). Los autores también agradecen el apoyo brindado por la Universidad Autónoma Metropolitana (UAM) y la Universidad Autónoma del Estado de Hidalgo (UAEH).
Referencias
Agyirifo, D.S., Wamalwa, M., Otwe, E.P., Galyuon, I., Runo, S., Takrama, J. & Ngeranwa, J. (2019). Metagenomics analysis of cocoa bean fermentation microbiome identifying species diversity and putative functional capabilities. Heliyon, 5(7), e02170. https://doi.org/10.1016/j.heliyon.2019.e02170
Akacha, B.B., Michalak, M., Najar, B., Venturi, F., Taglieri, I., Kačániová, M., Ben Saad, R., Mnif, W., Garzoli, S. & Ben Hsouna, A. (2023). Recent Advances in the Incorporation of Polysaccharides with Antioxidant and Antibacterial Functions to Preserve the Quality and Shelf Life of Meat Products. Foods, 12(8), 1647. https://doi.org/10.3390/foods12081647
Allali, I., Arnold, J.W., Roach, J., Cadenas, M.B., Butz, N., Hassan, H.M., Koci, M., Ballou, A., Mendoza, M., Ali, R. & Azcarate-Peril, M. A. (2017). A comparison of sequencing platforms and bioinformatics pipelines for compositional analysis of the gut microbiome. BMC Microbiology, 17(1). https://doi.org/10.1186/s12866-017-1101-8
Arango-Argoty, G., Singh, G., Heath, L.S., Pruden, A., Xiao, W. & Zhang, L. (2016). MetaStorm: A Public Resource for Customizable Metagenomics Annotation. PLOS ONE, 11(9), e0162442. https://doi.org/10.1371/journal.pone.0162442
Ban, S., Cheng, W., Wang, X., Niu, J., Wu, Q. & Xu, Y. (2024). Predicting the final metabolic profile based on the succession-related microbiota during spontaneous fermentation of the starter for Chinese liquor making. mSystems, 9(2). https://doi.org/10.1128/msystems.00586-23
Beck, K.L., Haiminen, N., Agarwal, A., Carrieri, A.P., Madgwick, M., Kelly, J., Pylro, V., Kawas, B., Wiedmann, M. & Ganda, E. (2024). Development and evaluation of statistical and artificial intelligence approaches with microbial shotgun metagenomics data as an untargeted screening tool for use in food production. mSystems, 9(11). https://doi.org/10.1128/msystems.00840-24
Billington, C., Kingsbury, J. M., & Rivas, L. (2022). Metagenomics Approaches for Improving Food Safety: A Review. Journal of Food Protection, 85(3), 448-464. https://doi.org/10.4315/jfp-21-301
Bloomfield, S.J., Hildebrand, F., Zomer, A.L., Palau, R. & Mather, A.E. (2025). Ecological insights into the microbiology of food using metagenomics and its potential surveillance applications. Microbial Genomics, 11(1). https://doi.org/10.1099/mgen.0.001337
Bourdichon, F., Arias, E., Babuchowski, A., Bückle, A., Bello, F.D., Dubois, A., Fontana, A., Fritz, D., Kemperman, R., Laulund, S., McAuliffe, O., Miks, M.H., Papademas, P., Patrone, V., Sharma, D.K., Sliwinski, E., Stanton, C., Von Ah, U., Yao, S. & Morelli, L. (2021). The forgotten role of food cultures. FEMS Microbiology Letters, 368(14). https://doi.org/10.1093/femsle/fnab085
Buytaers, F.E., Saltykova, A., Denayer, S., Verhaegen, B., Vanneste, K., Roosens, N.H.C., Piérard, D., Marchal, K. & De Keersmaecker, S.C.J. (2021). Towards Real-Time and Affordable Strain-Level Metagenomics-Based Foodborne Outbreak Investigations Using Oxford Nanopore Sequencing Technologies. Frontiers in Microbiology, 12. https://doi.org/10.3389/fmicb.2021.738284
Buytaers, F.E., Saltykova, A., Mattheus, W., Verhaegen, B., Roosens, N.H. C., Vanneste, K., Laisnez, V., Hammami, N., Pochet, B., Cantaert, V., Marchal, K., Denayer, S. & De Keersmaecker, S.C.J. (2021). Application of a strain-level shotgun metagenomics approach on food samples: Resolution of the source of a Salmonella food-borne outbreak. Microbial Genomics, 7(4). https://doi.org/10.1099/mgen.0.000547
Calabrese, F.M., Ameur, H., Nikoloudaki, O., Celano, G., Vacca, M., Junior, W. Jfl., Manzari, C., Vertè, F., Di Cagno, R., Pesole, G., De Angelis, M. & Gobbetti, M. (2022). Metabolic framework of spontaneous and synthetic sourdough metacommunities to reveal microbial players responsible for resilience and performance. Microbiome, 10(1). https://doi.org/10.1186/s40168-022-01301-3
Chileshe, J., Van Den Heuvel, J., Handema, R., Zwaan, B. J., Talsma, E. F. & Schoustra, S. (2020). Nutritional Composition and Microbial Communities of Two Non-alcoholic Traditional Fermented Beverages from Zambia: A Study of Mabisi and Munkoyo. Nutrients, 12(6), 1628. https://doi.org/10.3390/nu12061628
Chiriac, L.S. & Murariu, D. (2021). APPLICATION OF METAGENOMICS IN ECOLOGY: A BRIEF OVERVIEW. Current Trends in Natural Sciences, 10(19), 40-47. https://doi.org/10.47068/ctns.2021.v10i19.005
Coughlan, L.M., Cotter, P.D., Hill, C. & Alvarez-Ordóñez, A. (2015). Biotechnological applications of functional metagenomics in the food and pharmaceutical industries. Frontiers in Microbiology, 6. https://doi.org/10.3389/fmicb.2015.00672
Das, R., Tamang, B., Najar, I.N., Thakur, N. & Mondal, K. (2023). First report on metagenomics and their predictive functional analysis of fermented bamboo shoot food of Tripura, North East India. Frontiers in Microbiology, 14. https://doi.org/10.3389/fmicb.2023.1158411
De Filippis, F., La Storia, A., Villani, F. & Ercolini, D. (2019). Strain-Level Diversity Analysis of Pseudomonas fragi after In Situ Pangenome Reconstruction Shows Distinctive Spoilage-Associated Metabolic Traits Clearly Selected by Different Storage Conditions. Applied and Environmental Microbiology, 85(1). https://doi.org/10.1128/aem.02212-18
DeCastro, M.E., Rodríguez-Belmonte, E. & González-Siso, M.I. (2016). Metagenomics of Thermophiles with a Focus on Discovery of Novel Thermozymes. Frontiers in Microbiology, 7. https://doi.org/10.3389/fmicb.2016.01521
Dioso, C.M., Vital, P., Arellano, K., Park, H., Todorov, S.D., Ji, Y. & Holzapfel, W. (2020). Do Your Kids Get What You Paid for? Evaluation of Commercially Available Probiotic Products Intended for Children in the Republic of the Philippines and the Republic of Korea. Foods, 9(9), 1229. https://doi.org/10.3390/foods9091229
Du, R., Xiong, W., Xu, L., Xu, Y. & Wu, Q. (2023). Metagenomics reveals the habitat specificity of biosynthetic potential of secondary metabolites in global food fermentations. Microbiome, 11(1). https://doi.org/10.1186/s40168-023-01536-8
Dutta, B., Lahiri, D., Nag, M., Abukhader, R., Sarkar, T., Pati, S., Upadhye, V., Pandit, S., Amin, M. F. M., Al Tawaha, A. R. M. S., Kumar, M., & Ray, R. R. (2022). Multi-Omics Approach in Amelioration of Food Products. Frontiers in Microbiology, 13. https://doi.org/10.3389/fmicb.2022.955683
Eckstrom, K. & Barlow, J. W. (2019). Resistome metagenomics from plate to farm: The resistome and microbial composition during food waste feeding and composting on a Vermont poultry farm. PLOS ONE, 14(11), e0219807. https://doi.org/10.1371/journal.pone.0219807
Efendi, R., Restuhadi, F., Hasibuan, A.I.R.S., Lestari, M.I. & Rossi, E. (2024). Effect of adding bacteriocin from Lactobacillus pentosus strain MIL 195 on the quality of chicken sausage as an alternative natural preservative. BIO Web of Conferences, 99, 02023. https://doi.org/10.1051/bioconf/20249902023
Fan, J., Qu, G., Wang, D., Chen, J., Du, G. & Fang, F. (2023). Synergistic Fermentation with Functional Microorganisms Improves Safety and Quality of Traditional Chinese Fermented Foods. Foods, 12(15), 2892. https://doi.org/10.3390/foods12152892
Ferravante, C., Memoli, D., Palumbo, D., Ciaramella, P., Di Loria, A., D’Agostino, Y., Nassa, G., Rizzo, F., Tarallo, R., Weisz, A., & Giurato, G. (2021). HOME-BIO (sHOtgun MEtagenomic analysis of BIOlogical entities): A specific and comprehensive pipeline for metagenomic shotgun sequencing data analysis. BMC Bioinformatics, 22(S7). https://doi.org/10.1186/s12859-021-04004-y
Gautam, A., Poopalarajah, R., Ahmad, A.R., Rana, B., Denekew, T.W., Anh, N., Utenova, L., Kunwar, Y.S., Bhandari, N. N. & Jha, A.R. (2024). Ecological factors that drive microbial communities in culturally diverse fermented foods. Cold Spring Harbor Laboratory. https://doi.org/10.1101/2024.08.20.608727
Grützke, J., Gwida, M., Deneke, C., Brendebach, H., Projahn, M., Schattschneider, A., Hofreuter, D., El-Ashker, M., Malorny, B., & Al Dahouk, S. (2021). Direct identification and molecular characterization of zoonotic hazards in raw milk by metagenomics using Brucella as a model pathogen. Microbial Genomics, 7(5). https://doi.org/10.1099/mgen.0.000552
Grützke, J., Malorny, B., Hammerl, J.A., Busch, A., Tausch, S.H., Tomaso, H. & Deneke, C. (2019). Fishing in the Soup – Pathogen Detection in Food Safety Using Metabarcoding and Metagenomic Sequencing. Frontiers in Microbiology, 10. https://doi.org/10.3389/fmicb.2019.01805
Han, D., Yang, Y., Guo, Z., Chen, K., Dai, S., Zhu, Y., Wang, Y., Yu, Z., Wang, K., Liu, P., Rong, C. & Yu, Y. (2024). Metagenomics profiling of the microbial community and functional differences in solid-state fermentation vinegar starter (seed Pei) from different Chinese regions. Frontiers in Microbiology, 15. https://doi.org/10.3389/fmicb.2024.1389737
Hassan, M., Essam, T., & Megahed, S. (2018). Illumina sequencing and assessment of new cost-efficient protocol for metagenomic-DNA extraction from environmental water samples. Brazilian Journal of Microbiology, 49, 1-8. https://doi.org/10.1016/j.bjm.2018.03.002
Hu, W., Cai, W., Zheng, Z., Liu, Y., Luo, C., Xue, F. & Li, D. (2022). Study on the chemical compositions and microbial communities of cigar tobacco leaves fermented with exogenous additive. Scientific Reports, 12(1). https://doi.org/10.1038/s41598-022-23419-y
Huang, A.D., Luo, C., Pena-Gonzalez, A., Weigand, M.R., Tarr, C.L. & Konstantinidis, K.T. (2017). Metagenomics of Two Severe Foodborne Outbreaks Provides Diagnostic Signatures and Signs of Coinfection Not Attainable by Traditional Methods. Applied and Environmental Microbiology, 83(3). https://doi.org/10.1128/aem.02577-16
Hyeon, J.Y., Li, S., Mann, D. A., Zhang, S., Li, Z., Chen, Y. & Deng, X. (2018). Quasimetagenomics-Based and Real-Time-Sequencing-Aided Detection and Subtyping of Salmonella enterica from Food Samples. Applied and Environmental Microbiology, 84(4). https://doi.org/10.1128/aem.02340-17
Kalantar, K.L., Carvalho, T., De Bourcy, C.F.A., Dimitrov, B., Dingle, G., Egger, R., Han, J., Holmes, O.B., Juan, Y.-F., King, R., Kislyuk, A., Lin, M.F., Mariano, M., Morse, T., Reynoso, L.V., Cruz, D.R., Sheu, J., Tang, J., Wang, J., … DeRisi, J. L. (2020). IDseq—An open source cloud-based pipeline and analysis service for metagenomic pathogen detection and monitoring. GigaScience, 9(10). https://doi.org/10.1093/gigascience/giaa111
Koutsoumanis, K., Allende, A., Alvarez‐Ordóñez, A., Bolton, D., Bover‐Cid, S., Chemaly, M., Davies, R., De Cesare, A., Hilbert, F., Lindqvist, R., Nauta, M., Peixe, L., Ru, G., Simmons, M., Skandamis, P., Suffredini, E., Jenkins, C., Malorny, B., Ribeiro Duarte, A. S., … Herman, L. (2019). Whole genome sequencing and metagenomics for outbreak investigation, source attribution and risk assessment of food‐borne microorganisms. EFSA Journal, 17(12). https://doi.org/10.2903/j.efsa.2019.5898
Kovac, J. (2019). Precision Food Safety: A Paradigm Shift in Detection and Control of Foodborne Pathogens. mSystems, 4(3). https://doi.org/10.1128/msystems.00164-19
Lee, A.W.T., Ng, I.C.F., Wong, E.Y.K., Wong, I.T.F., Sze, R.P.P., Chan, K.Y., So, T.Y., Zhang, Z., Fung, S.K.Y., Wong, S.C.Y., Tam, W.Y., Lao, H.Y., Lee, L.K., Leung, J.S.L., Chan, C.T.M., Ng, T.T.L., Chow, F.W.N., Leung, P.H.M. & Siu, G.K.H. (2023). Comprehensive Identification of Pathogenic Microbes and Antimicrobial Resistance Genes in Food Products Using Nanopore Sequencing-Based Metagenomics. Cold Spring Harbor Laboratory. https://doi.org/10.1101/2023.10.15.562131
Leeuwendaal, N.K., Stanton, C., O’Toole, P.W. & Beresford, T.P. (2022). Fermented Foods, Health and the Gut Microbiome. Nutrients, 14(7), 1527. https://doi.org/10.3390/nu14071527
Lin, H.H., & Liao, Y.C. (2016). Accurate binning of metagenomic contigs via automated clustering sequences using information of genomic signatures and marker genes. Scientific Reports, 6(1). https://doi.org/10.1038/srep24175
Liu, S., Zhang, Z.F., Mao, J., Zhou, Z., Zhang, J., Shen, C., Wang, S., Marco, M.L. & Mao, J. (2023). Integrated meta-omics approaches reveal Saccharopolyspora as the core functional genus in huangjiu fermentations. Npj Biofilms and Microbiomes, 9(1). https://doi.org/10.1038/s41522-023-00432-1
Lugli, G.A., Longhi, G., Alessandri, G., Mancabelli, L., Tarracchini, C., Fontana, F., Turroni, F., Milani, C., Di Pierro, F., Van Sinderen, D. & Ventura, M. (2022). The Probiotic Identity Card: A Novel “Probiogenomics” Approach to Investigate Probiotic Supplements. Frontiers in Microbiology, 12. https://doi.org/10.3389/fmicb.2021.790881
Mazzantini, D., Celandroni, F., Calvigioni, M., Panattoni, A., Labella, R. & Ghelardi, E. (2021). Microbiological Quality and Resistance to an Artificial Gut Environment of Two Probiotic Formulations. Foods, 10(11), 2781. https://doi.org/10.3390/foods10112781
Miao, Z., Bai, Y., Wang, X., Han, C., Wang, B., Li, Z., Sun, J., Zheng, F., Zhang, Y. & Sun, B. (2023). Unravelling Metabolic Heterogeneity of Chinese Baijiu Fermentation in Age-Gradient Vessels. Foods, 12(18), 3425. https://doi.org/10.3390/foods12183425
Miller, I.J., Rees, E.R., Ross, J., Miller, I., Baxa, J., Lopera, J., Kerby, R.L., Rey, F.E. & Kwan, J.C. (2018). Autometa: Automated extraction of microbial genomes from individual shotgun metagenomes. Cold Spring Harbor Laboratory. https://doi.org/10.1101/251462
Munekata, P.E.S., Pateiro, M., Zhang, W., Domínguez, R., Xing, L., Fierro, E.M. & Lorenzo, J.M. (2020). Autochthonous Probiotics in Meat Products: Selection, Identification, and Their Use as Starter Culture. Microorganisms, 8(11), 1833. https://doi.org/10.3390/microorganisms8111833
Muñoz-Martinez, T.I., Rodríguez-Hernández, B., Rodríguez-Montaño, M., Alfau, J., Reyes, C., Fernandez, Y., Ramos, R.T., De Los Santos, E.F.F. & Maroto-Martín, L.O. (2025). Unlocking the Hidden Microbiome of Food: The Role of Metagenomics in Analyzing Fresh Produce, Poultry, and Meat. Applied Microbiology, 5(1), 26. https://doi.org/10.3390/applmicrobiol5010026
Oliveira, J.S., Araújo, W., Lopes Sales, A.I., Brito Guerra, A.D., Silva Araújo, S.C.D., De Vasconcelos, A.T.R., Agnez-Lima, L.F. & Freitas, A.T. (2015). BioSurfDB: Knowledge and algorithms to support biosurfactants and biodegradation studies. Database, 2015. https://doi.org/10.1093/database/bav033
Ottesen, A., Kocurek, B., Reed, E., Commichaux, S., Mammel, M., Ramachandran, P., McDermott, P., Flannery, B.M. & Strain, E. (2024). Paired metagenomic and chemical evaluation of aflatoxin-contaminated dog kibble. Frontiers in Veterinary Science, 11. https://doi.org/10.3389/fvets.2024.1374839
Pangastuti, A., Alfisah, R.K., Istiana, N.I., Sari, S.L.A., Setyaningsih, R., Susilowati, A. & Purwoko, T. (2019). Metagenomic analysis of microbial community in over-fermented tempeh. Biodiversitas Journal of Biological Diversity, 20(4), 1106-1114. https://doi.org/10.13057/biodiv/d200423
Pant, I. & Shashidhar, R. (2023). Inter-species competition of surface bacterial flora of pomegranate and their role in spoilage. World Journal of Microbiology and Biotechnology, 39(10). https://doi.org/10.1007/s11274-023-03709-x
Parras-Moltó, M. & Aguirre De Cárcer, D. (2020). A comprehensive human minimal gut metagenome extends the host’s metabolic potential. Microbial Genomics, 6(11). https://doi.org/10.1099/mgen.0.000466
Parrello, B., Butler, R., Chlenski, P., Pusch, G.D. & Overbeek, R. (2021). Supervised extraction of near-complete genomes from metagenomic samples: A new service in PATRIC. PLOS ONE, 16(4), e0250092. https://doi.org/10.1371/journal.pone.0250092
Pasolli, E., Asnicar, F., Manara, S., Zolfo, M., Karcher, N., Armanini, F., Beghini, F., Manghi, P., Tett, A., Ghensi, P., Collado, M.C., Rice, B.L., DuLong, C., Morgan, X.C., Golden, C.D., Quince, C., Huttenhower, C. & Segata, N. (2019). Extensive Unexplored Human Microbiome Diversity Revealed by Over 150,000 Genomes from Metagenomes Spanning Age, Geography, and Lifestyle. Cell, 176(3), 649-662.e20. https://doi.org/10.1016/j.cell.2019.01.001
Pasolli, E., De Filippis, F., Mauriello, I.E., Cumbo, F., Walsh, A.M., Leech, J., Cotter, P.D., Segata, N. & Ercolini, D. (2020). Large-scale genome-wide analysis links lactic acid bacteria from food with the gut microbiome. Nature Communications, 11(1). https://doi.org/10.1038/s41467-020-16438-8
Phiri, S., Schoustra, S. E., Van Den Heuvel, J., Smid, E. J., Shindano, J., & Linnemann, A. (2019). Fermented cereal-based Munkoyo beverage: Processing practices, microbial diversity and aroma compounds. PLOS ONE, 14(10), e0223501. https://doi.org/10.1371/journal.pone.0223501
Pineda-Quiroga, C., Borda-Molina, D., Chaves-Moreno, D., Ruiz, R., Atxaerandio, R., Camarinha-Silva, A. & García-Rodríguez, A. (2019). Microbial and Functional Profile of the Ceca from Laying Hens Affected by Feeding Prebiotics, Probiotics, and Synbiotics. Microorganisms, 7(5), 123. https://doi.org/10.3390/microorganisms7050123
Qayyum, H., Ali, A. & Rehman Kayani, M.U. (2024). EfficientDe NovoAssembly and Recovery of Microbial Genomes from Complex Metagenomes Using a Reduced Set ofk-mers. Cold Spring Harbor Laboratory. https://doi.org/10.1101/2024.06.08.598064
Qiao, H., Chen, L., Yang, J., Zhi, W., Chen, R., Lu, T., Tan, H. & Sheng, Z. (2022). Effect of Lactic Acid Bacteria on Bacterial Community Structure and Characteristics of Sugarcane Juice. Foods, 11(19), 3134. https://doi.org/10.3390/foods11193134
Quek, J.J.W., Wong, J.L., Tan, J.L., Yeo, C.C. & Saw, S.H. (2025). Integrating Metagenomic and Culture-Based Techniques to Detect Foodborne Pathogens and Antimicrobial Resistance Genes in Malaysian Produce. Foods, 14(3), 352. https://doi.org/10.3390/foods14030352
Raghuvanshi, R., Grayson, A.G., Schena, I., Amanze, O., Suwintono, K. & Quinn, R.A. (2019). Microbial Transformations of Organically Fermented Foods. Metabolites, 9(8), 165. https://doi.org/10.3390/metabo9080165
Ramiro-Garcia, J., Hermes, G.D.A., Giatsis, C., Sipkema, D., Zoetendal, E.G., Schaap, P.J. & Smidt, H. (2018). NG-Tax, a highly accurate and validated pipeline for analysis of 16S rRNA amplicons from complex biomes. F1000Research, 5, 1791. https://doi.org/10.12688/f1000research.9227.2
Rasheed, H.A., Tuoheti, T., Li, Z., Tekliye, M., Zhang, Y. & Dong, M. (2021). Effect of Novel Bacteriocinogenic Lactobacillus fermentum BZ532 on Microbiological Shelf-Life and Physicochemical and Organoleptic Properties of Fresh Home-Made Bozai. Foods, 10(9), 2120. https://doi.org/10.3390/foods10092120
Rintala, A., Pietilä, S., Munukka, E., Eerola, E., Pursiheimo, J.P., Laiho, A., Pekkala, S. & Huovinen, P. (2017). Gut Microbiota Analysis Results Are Highly Dependent on the 16S rRNA Gene Target Region, Whereas the Impact of DNA Extraction Is Minor. Journal of Biomolecular Techniques : JBT, 28(1), 19-30. https://doi.org/10.7171/jbt.17-2801-003
Rosales-Valdívia, B.S., García-Curiel, L., Pérez-Flores, J.G., Contreras-López, E., Pérez-Escalante, E. & García-Mora, C. (2024). Influencia de la fermentación del cacao y del uso de cultivos iniciadores sobre las características organolépticas del chocolate: Un análisis integral. Pädi Boletín Científico de Ciencias Básicas e Ingenierías del ICBI, 12(23), 31-43. https://doi.org/10.29057/icbi.v12i23.12047
Rus‐Fernández, P. & Fuentes, A. (2025). Fermentation starters and bacteriocins as biocontrol strategies for table olives preservation: A mini‐review. Journal of the Science of Food and Agriculture, 105(7), 3550-3556. https://doi.org/10.1002/jsfa.13874
Saltykova, A., Buytaers, F.E., Denayer, S., Verhaegen, B., Piérard, D., Roosens, N.H.C., Marchal, K. & De Keersmaecker, S. C.J. (2020). Strain-Level Metagenomic Data Analysis of Enriched In Vitro and In Silico Spiked Food Samples: Paving the Way towards a Culture-Free Foodborne Outbreak Investigation Using STEC as a Case Study. International Journal of Molecular Sciences, 21(16), 5688. https://doi.org/10.3390/ijms21165688
Sanico, T. & Medina, P. (2021). Metagenomic Characterization of the Culturable Bacterial Community Structure of Tapuy, a Philippine Indigenous Rice Wine, Reveals Significant Presence of Potential Probiotic Bacteria. Acta Medica Philippina. https://doi.org/10.47895/amp.vi0.3232
Santiago-Rodriguez, T.M., Cano, R. & Jiménez-Flores, R. (2016). Potential applications of metagenomics to assess the biological effects of food structure and function. Food & Function, 7(10), 4160-4169. https://doi.org/10.1039/c6fo00317f
Sedlar, K., Kupkova, K., & Provaznik, I. (2017). Bioinformatics strategies for taxonomy independent binning and visualization of sequences in shotgun metagenomics. Computational and Structural Biotechnology Journal, 15, 48-55. https://doi.org/10.1016/j.csbj.2016.11.005
Shamim, K., Sharma, J. & Dubey, S.K. (2017). Rapid and efficient method to extract metagenomic DNA from estuarine sediments. 3 Biotech, 7(3). https://doi.org/10.1007/s13205-017-0846-y
Sim, K.H., Ho, J., Lim, J.Q., Chan, S.H., Li, A. & Chng, K.R. (2024). A metagenomics-based workflow for the detection and genomic characterization of GBS in raw freshwater fish. Microbiology Spectrum, 12(6). https://doi.org/10.1128/spectrum.03276-23
Srinivas, M., O’Sullivan, O., Cotter, P.D., Sinderen, D.V. & Kenny, J.G. (2022). The Application of Metagenomics to Study Microbial Communities and Develop Desirable Traits in Fermented Foods. Foods, 11(20), 3297. https://doi.org/10.3390/foods11203297
Stewart, R.D., Auffret, M.D., Warr, A., Wiser, A.H., Press, M.O., Langford, K.W., Liachko, I., Snelling, T.J., Dewhurst, R.J., Walker, A.W., Roehe, R. & Watson, M. (2018). Assembly of 913 microbial genomes from metagenomic sequencing of the cow rumen. Nature Communications, 9(1). https://doi.org/10.1038/s41467-018-03317-6
Sumarmono, J., Kusuma, R.J., Rahayu, N., Sukarno, A.S. & Wulansari, P.D. (2023). Metagenomic analysis of the microbial community in kefir grains from different milk sources. Biodiversitas Journal of Biological Diversity, 24(10). https://doi.org/10.13057/biodiv/d241011
Sun, H., Wang, P. & Li, Y. (2023). An integrated microbiome project for charactering microbial diversity in classroom based on virtual simulation experiments. Biochemistry and Molecular Biology Education, 51(2), 171-179. https://doi.org/10.1002/bmb.21706
Tamames, J. & Puente-Sánchez, F. (2019). SqueezeMeta, A Highly Portable, Fully Automatic Metagenomic Analysis Pipeline. Frontiers in Microbiology, 9. https://doi.org/10.3389/fmicb.2018.03349
Tamang, J.P., Shin, D.H., Jung, S.J. & Chae, S.W. (2016). Functional Properties of Microorganisms in Fermented Foods. Frontiers in Microbiology, 7. https://doi.org/10.3389/fmicb.2016.00578
Taylor, B.C., Lejzerowicz, F., Poirel, M., Shaffer, J. P., Jiang, L., Aksenov, A., Litwin, N., Humphrey, G., Martino, C., Miller-Montgomery, S., Dorrestein, P.C., Veiga, P., Song, S. J., McDonald, D., Derrien, M. & Knight, R. (2020). Consumption of Fermented Foods Is Associated with Systematic Differences in the Gut Microbiome and Metabolome. mSystems, 5(2). https://doi.org/10.1128/msystems.00901-19
Tennant, R.K., Sambles, C.M., Diffey, G.E., Moore, K.A. & Love, J. (2017). Metagenomic Analysis of Silage. Journal of Visualized Experiments, 119. https://doi.org/10.3791/54936
Tourlousse, D.M., Narita, K., Miura, T., Sakamoto, M., Ohashi, A., Shiina, K., Matsuda, M., Miura, D., Shimamura, M., Ohyama, Y., Yamazoe, A., Uchino, Y., Kameyama, K., Arioka, S., Kataoka, J., Hisada, T., Fujii, K., Takahashi, S., Kuroiwa, M., … Terauchi, J. (2021). Validation and standardization of DNA extraction and library construction methods for metagenomics-based human fecal microbiome measurements. Microbiome, 9(1), 95. https://doi.org/10.1186/s40168-021-01048-3
Tsafrakidou, P., Michaelidou, A.M. & G. Biliaderis, C. (2020). Fermented Cereal-based Products: Nutritional Aspects, Possible Impact on Gut Microbiota and Health Implications. Foods, 9(6), 734. https://doi.org/10.3390/foods9060734
Verma, S.K., Singh, H., & Sharma, P.C. (2017). An improved method suitable for isolation of high-quality metagenomic DNA from diverse soils. 3 Biotech, 7(3). https://doi.org/10.1007/s13205-017-0847-x
Wagner, E., Fagerlund, A., Langsrud, S., Møretrø, T., Jensen, M.R. & Moen, B. (2021). Surveillance of Listeria monocytogenes: Early Detection, Population Dynamics, and Quasimetagenomic Sequencing during Selective Enrichment. Applied and Environmental Microbiology, 87(24). https://doi.org/10.1128/aem.01774-21
Walsh, A.M., Macori, G., Kilcawley, K.N. & Cotter, P.D. (2020). Meta-analysis of cheese microbiomes highlights contributions to multiple aspects of quality. Nature Food, 1(8), 500-510. https://doi.org/10.1038/s43016-020-0129-3
Wang, J., Schamp, C.N., Hudson, L.K., Chaggar, H.K., Bryan, D.W., Garman, K.N., Radosevich, M. & Denes, T.G. (2025). Whole-genome sequencing and metagenomics reveal diversity and prevalence of Listeria spp. From soil in the Nantahala National Forest. Microbiology Spectrum, 13(1). https://doi.org/10.1128/spectrum.01712-24
Whon, T.W., Ahn, S.W., Yang, S., Kim, J.Y., Kim, Y.B., Kim, Y., Hong, J.M., Jung, H., Choi, Y.E., Lee, S.H. & Roh, S.W. (2021). ODFM, an omics data resource from microorganisms associated with fermented foods. Scientific Data, 8(1). https://doi.org/10.1038/s41597-021-00895-x
Wikandari, R., Kinanti, D. A., Permatasari, R.D., Rahmaningtyas, N.L., Chairunisa, N.R., Sardjono, Hellwig, C. & Taherzadeh, M.J. (2021). Correlations between the Chemical, Microbiological Characteristics and Sensory Profile of Fungal Fermented Food. Fermentation, 7(4), 261. https://doi.org/10.3390/fermentation7040261
Yasir, M., Alkhaldy, A.A., Soliman, S.A., Turkistani, S.A. & Azhar, E.I. (2023). Metagenomic Insights into the Microbiome and Resistance Genes of Traditional Fermented Foods in Arabia. Foods, 12(18), 3342. https://doi.org/10.3390/foods12183342
Yu, W., Guo, J., Liu, Y., Xue, X., Wang, X., Wei, L. & Ma, J. (2023). Potential Impact of Combined Inhibition by Bacteriocins and Chemical Substances of Foodborne Pathogenic and Spoilage Bacteria: A Review. Foods, 12(16), 3128. https://doi.org/10.3390/foods12163128
Yue, Y., Huang, H., Qi, Z., Dou, H.M., Liu, X.Y., Han, T.F., Chen, Y., Song, X.J., Zhang, Y.H. & Tu, J. (2020). Evaluating metagenomics tools for genome binning with real metagenomic datasets and CAMI datasets. BMC Bioinformatics, 21(1). https://doi.org/10.1186/s12859-020-03667-3
Zabat, M.A., Sano, W.H., Wurster, J.I., Cabral, D.J. & Belenky, P. (2018). Microbial Community Analysis of Sauerkraut Fermentation Reveals a Stable and Rapidly Established Community. Foods, 7(5), 77. https://doi.org/10.3390/foods7050077
Zhang, J., Wang, X., Huo, D., Li, W., Hu, Q., Xu, C., Liu, S. & Li, C. (2016). Metagenomic approach reveals microbial diversity and predictive microbial metabolic pathways in Yucha, a traditional Li fermented food. Scientific Reports, 6(1). https://doi.org/10.1038/srep32524
Zorrilla, F., Buric, F., Patil, K.R. & Zelezniak, A. (2021). metaGEM: Reconstruction of genome scale metabolic models directly from metagenomes. Nucleic Acids Research, 49(21), e126-e126. https://doi.org/10.1093/nar/gkab815
