Análisis morfológico y molecular de especies de bambú del género Guadua (Poaceae: Bambusoideae) procedentes de las regiones San Martín y Cajamarca, Perú
Morphological and molecular analysis of bamboo species of the genus Guadua (Poaceae: Bambusoideae) from San Martin and Cajamarca regions, Peru
Romina Cecilia Móstiga Rodríguez1, Bruno Germán Cano Rodríguez1, Lilia Rosario Quispe López1, Maricel Jadith Móstiga Rodríguez1*
1Universidad Nacional Agraria La Molina (UNALM), Facultad de Ciencias Forestales, Avenida Victor Marie N° 15024, Lima, Perú
*Autor de correspondencia. E-mail: mmostiga@lamolina.edu.pe
Recibido, 29/11/2018 Aceptado, 02/02/2019 DOI:10.25127/aps.20191.486
![]()
RESUMEN
Palabras claves: Guadua, madera bambú, marcadores moleculares, ISSR.
ABSTRACT
In the San Martín Region, there is confusion regarding the identification of three species of the genus Guadua. According to the distribution and morphological characteristics it is determined that "Kunth" from La Florida (province of San Miguel, Cajamarca Region) corresponds to Guadua angustifolia Kunth, "Marona" from Atumplaya (province of Moyobamba, San Martin Region) corresponds to Guadua lynnclarkiae Londoño and "Guayaquil" from the San Martín region is defined as Guadua sp. as it does not have definite taxonomic identification. Through morphological and molecular analysis, morphological variables and molecular markers that allow discrimination of the species under study are identified. The morphological analysis was carried out in Guadua sp. and Guadua lynnclarkiae and consisted of daily measurements of: height of the plant, distance between nodes, number of nodes, number of branches, number of leaves, color of leaves (light green or orange) and diameter, during one month. Molecular analysis was carried out in the three species studied, the DNA was extracted and then the amplification was performed using the ISSR technique. The ISSR primers used were: IS-5, SSR 15, SSR 16 and SSR 22. The variables morphological differences that allowed to differentiate Guadua sp. and Guadua lynnclarkiae were the "height" and "number of leaves". While, the IS-5 initiator allows to discriminate Guadua sp. from Guadua lynnclarkiae and Guadua angustifolia, the primers SSR 15, SSR 16 and SSR22 did not allow to differentiate the species under study.
Key words: Guadua, woody bamboo, molecular markers, ISSR.
I. INTRODUCCIÓN
La familia Poaceae comprende aproximadamente 11500 especies contenidas en 768 géneros, incluyendo a dos tribus de bambúes leñosos: Arundinarieae (31 géneros, 581 especies) y Bambuseae (73 géneros, 966 especies), siendo esta última tribu característica del neotrópico (Soreng et al., 2017). El bambú es una planta multipropósito, es decir, puede sustituir a la madera en muchos aspectos debido a sus culmos lignificados, y debido a su rápido crecimiento (Clark et al., 2015).
La subtribu Guaduinae (5 géneros, 53 especies) pertenece a la tribu Bambuseae, sus especies habitan en bosques tropicales secos o húmedos de baja a mediana elevación y forman extensos bosques mono-dominantes de Guadua en parte de la cuenca del Amazonas (Judziewicz et al. 1999). Guadua es principalmente un género de tierras bajas, pero algunas de sus especies se extienden hacia las colinas y los bosques montanos más bajos de los Andes (Clark, 2001).
Los bambúes son plantas complejas que pueden ser difíciles de identificar o clasificar, pero dada su importancia ecológica y económica, la identificación correcta es fundamental para su conservación y desarrollo (Clark et al., 2015). Esto se debe a que la mayoría de ellos tienen largos períodos vegetativos con una etapa reproductiva corta, tal es el caso del género Guadua, motivo por el cual la identificación de los bambúes leñosos actuales se basa principalmente en caracteres vegetativos (Botero-Cortés, 2003; Judziewicz et al. 1999, citado por Brea y Zucol, 2007).
Las especies nativas, al representar bienes asequibles y de disposición inmediata para el poblador local, constituyen un recurso potencialmente productivo y rentable. Sin embargo, existen vacíos de información y confusiones en cuanto a la identificación taxonómica, la ecología y las consideraciones para su manejo, como es el caso de las especies mencionadas en el presente estudio.
Debido a ello, el Círculo de Investigación en la Cadena de Valor del Bambú para el Desarrollo, Científico y Tecnológico (CIB) surge como una propuesta para la contribución del conocimiento del recurso forestal, dada su difundida importancia ambiental, social y económica.
Los rangos de acción del CIB están constituidos por dos macroregiones: la nor-oriental, constituida por las Regiones de Amazonas, Cajamarca y San Martín, y selva central, constituida por las Regiones de Pasco y Junín. En la macro región nor-oriental, durante los últimos años, se ha impulsado el desarrollo de la silvicultura del bambú desde viveros hasta plantaciones establecidas. Específicamente en la región San Martín, el Proyecto Especial Alto Mayo (PEAM) ha creado incentivos para el desarrollo de este importante recurso, promoviendo constantes capacitaciones a los pequeños productores, quienes actualmente conforman una asociación.
En la región San Martín, los pobladores identifican tres especies de Guadua en función a sus diferencias morfológicas y usos, estas especies son denominadas por nombre común como: “Kunth”, “Guayaquil” y “Marona”. Según las características morfológicas y distribución geográfica de la colección de las muestras, “Kunth” procedente de La Florida (provincia de San Miguel, Región Cajamarca) es identificada taxonómicamente como la especie Guadua angustifolia Kunth, “Marona” procedente de Atumplaya (provincia de Moyobamba, Región San Martín) es identificada como la especie Guadua lynnclarkiae Londoño y “Guayaquil” procedente de la Región San Martín, no tiene identificación taxonómica definida, por lo que será nombrada como Guadua sp. para los fines del presente estudio.
A nivel morfológico, en condiciones de vivero, se observa una marcada diferencia entre Guadua sp. “Guayaquil” y Guadua angustifolia “Kunth”: la primera genera entre 20 y 40 hijuelos[1] (Figura 1) con un periodo de madurez en campo de 2 a 3 años, mientras que la segunda genera entre 6 a 10 hijuelos y su periodo de madurez en campo es menor a 6 meses; sin embargo, para el caso de la “Guayaquil”, la capacidad de generar una gran cantidad de hijuelos se mantiene en campo con lo que su crecimiento en altura se retrasa y las actividades de manejo aumentan, a diferencia de la “Kunth”, que una vez en campo crece en altura en lugar de seguir generando hijuelos (Figura 2).

Figura 1. Mata de Guadua sp. “Guayaquil” con 40 hijuelos en promedio.

Figura 2. Plantación experimental del Proyecto Especial Alto Mayo (PEAM) de las especies Guadua angustifolia “Kunth” de 3 meses (A) y Guadua sp. “Guayaquil” de 10 meses (B) en el distrito de Calzada, bajo mismas condiciones de suelo.
En el distrito de Posic, provincia de Rioja, región San Martín, se puede constatar la presencia de plantaciones de bambú, las cuales cuentan con especies propias de la zona y de diferentes edades, encontrándose matas establecidas. Las plantaciones de Guadua angustifolia “Kunth” son fáciles de manejar al tener un número promedio de cañas por planta y crecer rápidamente en altura, las plantaciones de Guadua sp. “Guayaquil” son de alto mantenimiento por la cantidad excesiva de hijuelos, mientras que las de Guadua lynnclarkiae “Marona” son difíciles de manejar por la cantidad de espinas, el empalizado y el reducido número de cañas por planta (Figura 3).

Figura 3. Plantaciones en Posic, Rioja, San Martín: A, “Marona”. B, “Guayaquil” sin manejo. C, “Guayaquil” con manejo.
En cuanto al uso, el conocimiento empírico en construcción local indica que Guadua sp. “Guayaquil” es una caña más resistente que Guadua angustifolia “Kunth”, mientras que la Guadua lynnclarkiae “Marona” es vidriosa, es decir, propensa a rajarse. Se requiere de estudios a nivel anatómico y de propiedades físico-mecánicas que sustenten lo anterior (Tabla 1).
Tabla 1. Resumen de características entre especies
|
|
G. angustifolia “Kunth” |
Guadua sp. "Guayaquil" |
G. lynnclarkiae “Marona” |
|
Manejo en vivero |
3 meses |
3 meses |
2 meses |
|
Producción (3 meses) |
4-6 hijuelos |
25-30 hijuelos |
4-6 hijuelos |
|
Hojas |
Anchas y oblongas |
Anchas y oblongas |
Menudas |
|
Porte |
Alto |
Bajo |
Mediano |
|
Establecimiento definitivo |
6 meses |
3 años |
- |
|
Calidad de caña |
Alta |
Alta |
Baja |
Cabe resaltar que, según Londoño (2010) en el marco del proyecto “Promoción de la rehabilitación, manejo y uso sostenible de los bosques tropicales de bambú en la región noroccidental del Perú”, identifica tres biotipos para la que denomina Guadua aff. angustifolia. El nombre común de la Guadua aff. angustifolia Biotipo 1 Lamas es "Marona", el nombre común de la Guadua aff. angustifolia Biotipo 2 Aramango es "Guayaquil" o "Marona" y el nombre común de la Guadua aff. angustifolia Biotipo 3 Atumplaya es "Marona" (al rodal se le conoce como “Maronal”). Posteriormente, Londoño (2013) registró dos nuevas especies de Guadua para el Perú, las cuales fueron G. lynnclarkiae Londoño y G. takahashiae Londoño, las cuales se asume que son correspondientes a dos de los biotipos previamente mencionados; sin embargo, existe confusión en la región en torno a la identificación y determinación de estos especímenes.
Los marcadores moleculares tienen una serie de características favorables en comparación con los marcadores morfológicos, ya que permiten revelar un mayor número de polimorfismos, identifican también las mutaciones silenciosas y sinónimas escondidas en las proteínas, además no se encuentran influenciados por el ambiente o por interacciones genéticas complejas y son considerados en general caracteres neutrales, con muy poco o ningún valor adaptativo (Gonzále-Fortes, 2008) motivo por el cual la caracterización molecular es de gran importancia para diferenciar estas especies de bambú
Existen diferentes tipos de marcadores moleculares de los cuales se obtiene información de diferente naturaleza. Las dos categorías principales son: a) marcadores codominantes, como por ejemplo los marcadores microsatélites, que revelan las diferentes variantes de un alelo (llamados multi - alelos), pero analizan sólo un locus y se necesita información previa de las secuencias y b) los marcadores dominantes, como por ejemplo los AFLP (Amplified Fragment Length Polymorphism) y los ISSR (Inter Simple Sequence Repeat) que permiten la detección de la presencia o ausencia de un alelo por cada locus pero simultáneamente para un gran número de loci (multi locus). Estos multi locus detectados son individualmente menos informativos; sin embargo, los sistemas de marcadores moleculares dominantes alcanzan su poder estadístico al presentar numerosos loci di-alélicos del genoma entero. De esta forma, una técnica de marcadores moleculares de tipo dominante puede llegar a superar a una de marcadores codominantes en la discriminación de taxas y poblaciones (Oyanedel, 2010).
En este contexto, la presente investigación busca contribuir con la clasificación taxonómica de especies del género Guadua mediante la identificación de variables morfológicas y marcadores moleculares que permitan la discriminación de las especies en estudio.
II. MATERIALES Y MÉTODOS
Localización del sitio experimental
El material vegetal de Guadua sp. “Guayaquil” y Guadua lynnclarkiae “Marona” fue obtenido a partir de plantas provenientes de la Granja Ganadera de Calzada, perteneciente al Proyecto Especial Alto Mayo (PEAM), ubicada en la provincia de Moyobamba, Región San Martín lugar donde se desarrollan de manera natural, mientras que el material vegetal de Guadua angustifolia “Kunth” fue obtenido de las plantaciones ubicadas en La Florida, Cajamarca.
Este material fue traslado, aclimatado y propagado en las instalaciones del Vivero Forestal, perteneciente a la Facultad de Ciencias Forestales de la Universidad Nacional Agraria La Molina, ubicado en el distrito de La Molina, Lima, Perú donde se realizaron las evaluaciones morfológicas. Este vivero se localiza a 240 m.s.n.m., en las coordenadas siguientes: 12°05’ latitud sur y 76°56’ latitud oeste. Su clima corresponde, según las zonas de vida de Holdridge, a un desierto desecado-Subtropical (dd-S), el cual se caracteriza por tener una biotemperatura media anual máxima de 22,2°C y la media mínima de 17,9°C, así como un promedio máximo de precipitación total por año de 44 mm y un promedio mínimo de 2,2 mm (INRENA, 1995).
Por otro lado, la fase molecular se realizó en el Laboratorio de Marcadores Moleculares del Programa de Cereales y Granos Nativos de la Universidad Nacional Agraria La Molina.
Análisis morfológico
El análisis morfológico se realizó en plantas coetáneas y a iguales condiciones de Guadua sp. “Guayaquil” y Guadua lynnclarkiae “Marona”. Para la selección de los morfológicos se tomó en cuenta las características fenotípicas de sencilla identificación visual, tales como forma de hoja, pubescencia, color de fruto, entre otros. En todos los casos debe tratarse de caracteres de herencia monogénica y predecible según las leyes de Mendel (Simpson, 1997). Muchos de ellos se convierten en importantes descriptores a la hora de inscribir nuevas variedades (Levitus et al., 2010).
En este sentido, se realizaron mediciones de: altura de la planta, distancia entre nudos, número de nudos, número de ramificaciones, número de hojas, color de hojas (verde claras o de color anaranjado) y diámetro, durante evaluaciones diarias, en el periodo de un mes.
Cabe resaltar que Guadua angustifolia “Kunth” no formó parte del análisis morfológico al contar con plantas de edad diferente en comparación con las demás especies en estudio.
Análisis molecular
El análisis molecular consistió en el uso de marcadores moleculares, los cuales se definen como segmentos de ADN con una ubicación específica en un cromosoma (punto de referencia) cuya herencia puede seguirse en individuos de una población. La secuencia puede pertenecer a regiones codificantes o sin función conocida (Andersen y Lübberstedt, 2003; Levitus et al., 2010). Para Valadez y Kahl (2000) un marcador se refiere a cualquier molécula de proteína, ARN o ADN de tamaño o peso molecular conocido que sirve para monitorear o calibrar la separación de las mismas utilizando electroforesis o cromatografía, y un marcador genético como cualquier gen cuya expresión permite un efecto fenotípico que puede ser detectado fácilmente (por ejemplo, un gen que ocasiona resistencia para algún antibiótico).
Los diferentes tipos de marcadores se distinguen por su capacidad de detectar polimorfismos en loci únicos o múltiples y son de tipo dominante o codominante (Simpson, 1997).
Según lo expuesto, el presente análisis molecular se realizó mediante la lectura de marcadores moleculares del tipo dominante, con la técnica ISSR, utilizando los iniciadores IS-5, SSR 15, SSR 16 y SSR22. En este sentido, se realizó la extracción de ADN de tejidos vegetales utilizado el protocolo descrito por Doyle y Doyle (1987) a partir de hojas de Guadua lynnclarkiae “Marona”, Guadua sp. “Guayaquil” y Guadua angustifolia “Kunth” (Tabla 2).
Tabla 2. Primers o iniciadores que fueron usados en la investigación
|
Primer |
Motivos |
SEQUENCE 5' --> 3' |
|
IS-5 |
(AC)8TG |
ACACACACACACACACTG |
|
SSR15 |
CCC(GT)6 |
CCCGTGTGTGTGTGT |
|
SSR16 |
GSG(GT)6 |
GSGGTGTGTGTGTGT |
|
SSR22 |
SSWN(GACA)3 |
SSWNGACAGACAGACA |
La primera etapa del procedimiento consistió en utilizar maceración mecánica para romper las paredes y membranas celulares del tejido en presencia de nitrógeno líquido. Luego, en la segunda etapa, el tejido vegetal es suspendido en un tampón de extracción que contenga detergente, antioxidante, EDTA (Ethylenediaminetetraacetic acid, en español, ácido etilen diamino tetra acético) y el agente tamponante, con la finalidad de desnaturalizar las proteínas preservando el ADN de la acción de enzimas de degradación. Esta suspensión se somete a una temperatura entre 50 y 65 °C durante 15 a 60 minutos, para facilitar su solubilización y homogeneización.
En la tercera etapa, se somete la suspensión a una extracción con solvente orgánico, cloroformo-alcohol isoamílico, donde las fases acuosa y orgánica se separan mediante centrifugación, observándose que en la fase inferior son retenidos la mayoría de los lípidos, proteínas y polisacáridos, mientras que el ADN, ARN y algunos polisacáridos quedan retenidos en la fase acuosa (superior).
En la cuarta etapa, es adicionado un alcohol a la fase acuosa, donde se presencia que el ADN forma un precipitado visible, que cuando es sedimentado por centrifugación es llamado “pellet”. Luego, en la quinta etapa, después del lavado de alcohol, a este precipitado de ADN/ARN se lo diluye con agua milli-Q y una solución que contiene ARNsa para degradar el ARN presente, quedando así sólo el ADN genómico deseado (Ferreira y Grattapaglia, 1998).
Posteriormente, se procedió a observar la pureza, cantidad y calidad del ADN mediante un biofotómetro y una cámara electroforética; para después de ello, con resultados adecuados realizar la reacción de cadena de la polimerasa (PCR), el cual es un procedimiento in vitro para la síntesis y duplicación de secuencias específicas de ADN (Azofeifa-Delgado, 2006). La ADN polimerasa dependiente es una enzima que, en unas condiciones determinadas y en presencia de una pequeña cadena de ADN, que actúa como cebador, es capaz de producir millones de copias de determinados fragmentos del ADN. Estos fragmentos se separan posteriormente por peso molecular y conformación mediante técnicas electroforéticas (Rallo et al. 2002), en este caso a 200 voltios, realizándose luego el revelado con nitrato de plata. La lectura de la placa con gel poliacrilamida y nitrato de plata consta del conteo de bandas de izquierda a derecha de los individuos utilizados según cada marcador molecular y, a partir de este patrón de bandas específico, se diferencian individuos por especies.
Análisis de datos
Para el análisis de los datos morfológicos, se utilizaron técnicas multivariadas (Componentes principales, análisis de agrupamiento). Asimismo, para el análisis de caracterización molecular, los datos fueron compilados como presencia o ausencia de una banda (1 y 0, respectivamente). La carencia o duda de un dato se registró por 9 (denominación arbitraria). Luego, se procedió a calcular el coeficiente de similitud denominado Simple Matching Coefficient (SMS), el cual dará igual peso a ausencias (0,0) como a las presencias (1,1). La conformación de grupos generará un dendrograma que permitirá visualizar las distancias genéticas entre las entradas analizadas. Estos análisis se realizarán usando el programa PAST versión 2.17 (Hammer et al., 2001).
III. RESULTADOS Y DISCUSIÓN
Existen dos clases de marcadores genéticos: los morfológicos y los moleculares (Tanksley, 1983). La caracterización e identificación tradicional de variedades se ha basado en el empleo de caracteres morfológicos (Rallo et al., 2002). No obstante, según Azofeifa-Delgado (2006), el uso de marcadores morfológicos en las plantas tiene muchas limitantes, pues su expresión puede estar sujeta a factores ambientales o fenológicos. Por otro lado, gracias a los avances en la biología molecular, se han desarrollado métodos de identificación y caracterización basados en el uso de marcadores moleculares que superan, en la gran mayoría de los casos, las limitaciones de los métodos tradicionales. A partir de las características morfológicas evaluadas en las primeras fechas de crecimiento de Guadua sp. “Guayaquil” y Guadua lynnclarkiae “Marona”, se realizó el Análisis de Componentes Principales, observándose solapamiento entre ambas para luego diferenciarse en las tomas de datos finales (Figura 4). En este sentido, los componentes principales 1 (altura) y 2 (número de hojas) representan el 85% de la variabilidad en las variables.
Pérez-Galindo (2014) menciona la existencia de una correlación entre el diámetro internudo con las variables asociadas con la altura total del tallo, lo cual indicaría que las poblaciones de bambú con paredes de mayor grosor poseen tallos de mayor altura y diámetro aún más grande. Asimismo, considera que tanto la base y la altura de la hoja caulinar, como la anchura y la longitud de la lámina foliar soporta la hipótesis de que estas asociaciones están relacionadas con la adquisición y el almacenamiento de nutrientes indicados anteriormente. En este sentido, los componentes principales obtenidos, primer componente correspondiente a la variable altura y el segundo al número de hojas, serían aquellas a las que se debería de dar mayor importancia en el momento de toma de datos.
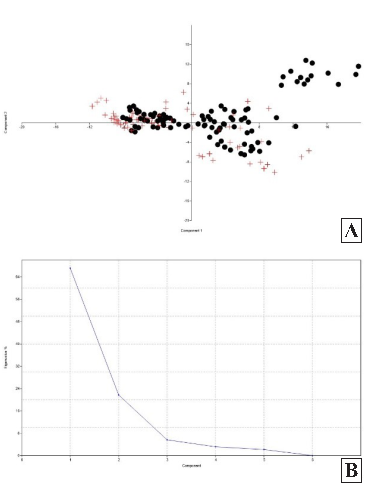
Figura 4. Análisis de Componentes Principales (ACP) de Guadua sp. “Guayaquil” y G. lynnclarkiae “Marona”. (A). ACP. (B). Línea de tendencia de variabilidad en las componentes principales.
En relación al análisis molecular, se procedió a realizar un dendrograma (Figura 5) que agrupa a los individuos evaluados según cada marcador. La información fue obtenida a partir de los datos provenientes de la lectura de la placa con gel poliacrilamida y nitrato de plata.
El PCR utilizado fue con sitio “objetivo específico”, y se utilizaron 4 primers, permitiendo amplificar nuestros microsatélites. Es importante mencionar que los marcadores de ADN se basan fundamentalmente en el análisis de las diferencias en pequeñas secuencias del ADN entre individuos (Azofeifa-Delgado, 2006).
Cervera et al. (2002) mencionan que, aunque los microsatélites permiten analizar sólo un locus por experimento, son bastante informativos ya que dejan diferenciar las variantes alélicas de los loci analizados y por lo tanto identificar grupos de ligamiento entre diferentes mapas genéticos. Estos marcadores suelen ser considerados ideales para el estudio de ligamiento genético en plantas y el mapeo físico, estudios poblacionales y la identificación de variedades (Ferreira y Grattapaglia, 1998); sin embargo, los SSR 15 y SSR 16 utilizados para el análisis no mostraron diferencias significativas entre los grupos, por lo que posiblemente su uso no aportaría información suficiente sobre la diferenciación entre estas especies de Guadua. Asimismo, el marcador molecular SSR22 no fue significativo para explicar el polimorfismo, debido a que la característica que representa estaría presente en todos los individuos, por lo cual no se le considera influyente para la toma de decisiones.
Por su parte, en la Figura 5(b) se puede observar cercanía entre Guadua lynnclarkiae “Marona” y Guadua angustifolia “Kunth”, que de Guadua lynnclarkiae “Marona” con Guadua sp. “Guayaquil”, siendo el marcador IS-5 aquel que permitió discriminar entre especies, por lo cual se deduce que posiblemente sería el mejor indicador para la el presente análisis.
En síntesis, el análisis con marcadores, así como el uso de caracteres morfológicos, aporta al conocimiento del género Guadua.
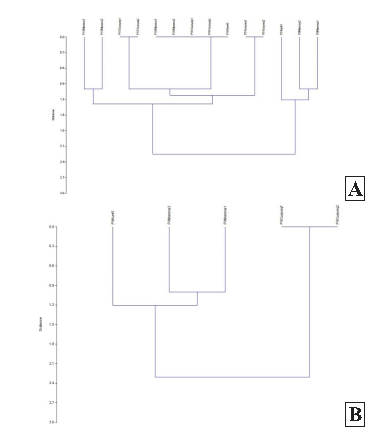
Figura 5. Dendrogramas del análisis molecular. (A) Dendrograma Marcador SSR15 (P15), Marcador IS-5 (P9), y Marcador SSR16 (P16). (B). Dendrograma Marcador IS-5 (P9).
IV. CONCLUSIONES
Las variables “altura” y “número de hojas” permiten diferenciar a nivel morfológico a las especies Guadua sp. “Guayaquil” y Guadua lynnclarkiae “Marona”.
El marcador molecular de Inter Secuencias IS-5 permite discriminar a Guadua sp. “Guayaquil” de Guadua lynnclarkiae “Marona” y Guadua angustifolia “Kunth”.
Los Microsatélites o Secuencias Simples Repetidas SSR 15, SSR 16 y SSR22 no permiten diferenciar a Guadua sp. “Guayaquil” de Guadua lynnclarkiae “Marona” y Guadua angustifolia “Kunth”.
V. AGRADECIMIENTOS
El presente estudio fue realizado gracias a la colaboración del Vivero Forestal de la Facultad de Ciencias Forestales y al Laboratorio de Marcadores Moleculares del Programa de Cereales y Granos Nativos de la Facultad de Agronomía, ambas entidades pertenecientes a la Universidad Nacional Agraria La Molina, por la disposición de sus instalaciones. De igual manera, se agradece a los alumnos de la Facultad de Ciencias de la carrera de Biología: Hernán Cunza Cáceres, Maura Isabel Fernández Chuquiyauri y Andrea Alicia Yacavilca Huarca, por sus aportes.
Este trabajo de investigación se realizó con el apoyo y financiamiento de FONDECYT-CONCYTEC, programa Ciencia Activa, del convenio N°174-2015 “Círculo de investigación para el Desarrollo de la Cadena de Valor del Bambú para el Desarrollo Científico y Tecnológico”.
VI. REFERENCIAS BIBLIOGRÁFICAS
Andersen, J. R., y T. Lübberstedt. 2003. “Functional markers in plants.” Trends in Plant Science 8 (11): 554-560.
Azofeifa-Delgado, Á. 2006. "Uso de marcadores moleculares en plantas; aplicaciones en frutales del trópico." Agronomía Mesoamericana 17 (2): 221-242.
Botero-Cortés, L. F. 2003. Reproducción de la Guadua angustifolia por el método de chusquines. Guayaquil (Ecuador): INBAR.
Brea, M., y A. F. Zucol. 2007. “Guadua zuloagae sp. nov., the first petrified bamboo culm record from the Ituzaingó Formation (Pliocene), Paraná Basin, Argentina”. Annals of Botany 100 (4): 711-723.
Cervera, M. T., J. A. Cabezas., y J. M. Martínez-Zapater. 2001. “Análisis genético de la vid: Una asignatura pendiente”. ACE: Revista de enología (12): 2.
Clark, L. G. 2001 “Diversification and endemism in Andean woody bamboos (Poaceae: Bambusoideae)”. Journal of the American Bamboo Society 15 (1): 14-19.
Clark, L. G., X. Londoño, y E. Ruiz-Sanchez. 2015. “Bamboo Taxonomy and Habitat”. En Bamboo. Liese, W., y M. Köhl (eds). Cham (Suiza): Springer International Publishing Switzerland
Doyle, J. J., y J. L. Doyle. 1987. “A rapid DNA isolation procedure for small quantities of fresh leaf tissue”. Phytochemical Bulletin 19 (1): 11-15.
Ferreira, M. E., y D. Grattapaglia. 1998. Introducción al uso de marcadores moleculares en el análisis genético. Brasilia (Brasil): EMBRAPA.
Gonzále-Fortes, G. M. 2008. Desarrollo de un mapa genético con marcadores AFLP y microsatélites en Rodaballo (Scophthalmus maximus L.). Tesis doctoral. Universidad de Santiago de Compostela. A Coruña (España).
Hammer, Ø., D. A. Harper, y P. D. Ryan. 2001. “Paleontological statistics software: package for education and data analysis”. Palaeontologia Electronica 4 (1): 1-9.
Judziewicz, E. J., L. G. Clark, X. Londoño, y M. J. Stern. 1999. American bamboos. Washington D.C. (USA): Smithsonian Institution Press.
Levitus, G., V. Echenique, C. Rubinstein, E. Hopp, L. Mroginski. 2010. Biotecnología y Mejoramiento Vegetal II. Buenos Aires (Argentina): Ediciones Instituto Nacional de Tecnología Agropecuaria.
Londoño, X. 2010. Identificación taxonómica de los bambúes de la región noroccidental del Perú. Lima (Perú): Perubambú.
Londoño, X. 2013. “Dos nuevas especies de Guadua para el Perú (Poaceae: Bambusoideae: Bambuseae: Guaduinae)”. Journal of the Botanical Research Institute of Texas 7 (1): 145-153.
Oyanedel, N. F. 2010. Análisis de la variabilidad genética de ejemplares de la especie vegetal Broussonetia papyrifera en la Polinesia: Evaluación mediante AFLP de un nuevo trazador de rutas migratorias humanas. Tesis de Maestría. Universidad de Chile. Santiago (Chile).
Pérez-Galindo, C. A. 2014. Caracterización genética de relictos de Guadua angustifolia, un ecosistema estratégico de la ecoregión Valle del Cauca mediante STR’s. Tesis Doctoral. Universidad Pablo de Olavide de Sevilla. Sevilla (España)
Rallo, P., A. Belaj, R. de la Rosa, y I. Trujillo. 2002. Marcadores moleculares. Córdoba (España): Caudal de Extremadura.
Simpson, J. 1997. “Amplified fragment length polymoorphisms.” Boletín de la Sociedad Botánica de México 60: 73-76.
Soreng, R. J., P. M. Peterson., K. Romaschenko., G. Davidse., J. K. Teisher., L. G. Clark., P. Barbera., L. J. Gillespie., y F. O. Zuloaga. 2017. “A worldwide phylogenetic classification of the Poaceae (Gramineae) II: An update and a comparison of two 2015 classifications”. Journal of Systematics and Evolution 55 (4): 259–290.
Tanksley, S. D. 1983. “Molecular markers in plant breeding”. Plant Molecular Biology Reporter 1 (1): 3-8.
Valadez, M. E., y Kahl, G. 2000. Huellas de ADN en genomas de plantas. Madrid (España): Mundi Prensa.